

发布时间:2021-08-16
单碱基编辑技术是近些年来基于CRISPR/Cas系统改造发展的新型基因编辑技术,它可以在不引入DNA双链断裂的情况下,精确地将DNA或者RNA中的一个碱基替换为另一个碱基。目前已经开发并得到广泛应用的碱基编辑器包括胞嘧啶碱基编辑器(cytosine base editor, CBE)以及腺嘌呤碱基编辑器(adenine base editor, ABE),但是这两种碱基编辑器只能实现将C·G碱基对替换为T·A碱基对(C→T),或者将A·T替换为G·C(A→G)。因此,CBE或者ABE只能修复由C>T或者A>G导致的遗传表型或者疾病,而对于其它类型的单碱基突变却束手无策。2020年,科研人员在CBE的基础上,研发了能够将胞嘧啶转换为鸟嘌呤的碱基编辑器(C-to-G base editor, CGBE)。但关于CGBE编辑器的研究仍处于初步阶段,对于其特异性、保真性以及编辑特点仍需进一步研究。David Liu实验室与其合作者对CGBEs系统进行改造与升级,构建了高效的CGBEs编辑器。为能够方便科研人员的日常研究,人工智能与基因编辑结合的越来越紧密,David Liu及Hyongbum Henry Kim等实验室分别已经建立了能够预测单碱基编辑器编辑效果的BE-Hive及DeepBE等深度学习模型。
孙怡迪研究组与左二伟研究组首先通过筛选不同物种来源的UNGs、密码子优化和全基因组及转录组范围测序,获得了可进行高效C到G碱基颠换以及高保真的OPTI-CGBEs。为了方便其他研究人员选择合适的C-to-G碱基编辑器以及高效预估编辑效率,研究人员建立了预测不同C-to-G碱基编辑器编辑效果的深度学习模型CGBE-SMART(图1)。CGBE-SMART结合了神经网络以及概率图模型,为每一个编辑位置独立训练一套参数来预测该位置上的编辑效率。模型使用了大小不同的卷积核,建立一组基础单元网络对编辑位置周围的碱基进行特征提取和效率预测。最终研究人员将不同基础单元网络的预测结果用一套习得的参数进行加权平均。模型以编辑位点附近的40bp作为输入,通过神经网络预测出guide RNA结合位置1至20的编辑效率并进一步利用贝叶斯网络预测不同编辑结果的占比(图1a)。研究人员将CGBE-SMART用在不同的CGBE编辑器的8个文库数据集上进行实验。在所有的8个数据集上,BE-SMART具有较高的预测准确性(图1b)。CGBE-SMART不仅能够准确预测C-to-G编辑效率,而且与之前的预测模型相比,在预测C-to-T编辑效果中也有更为出色的表现。
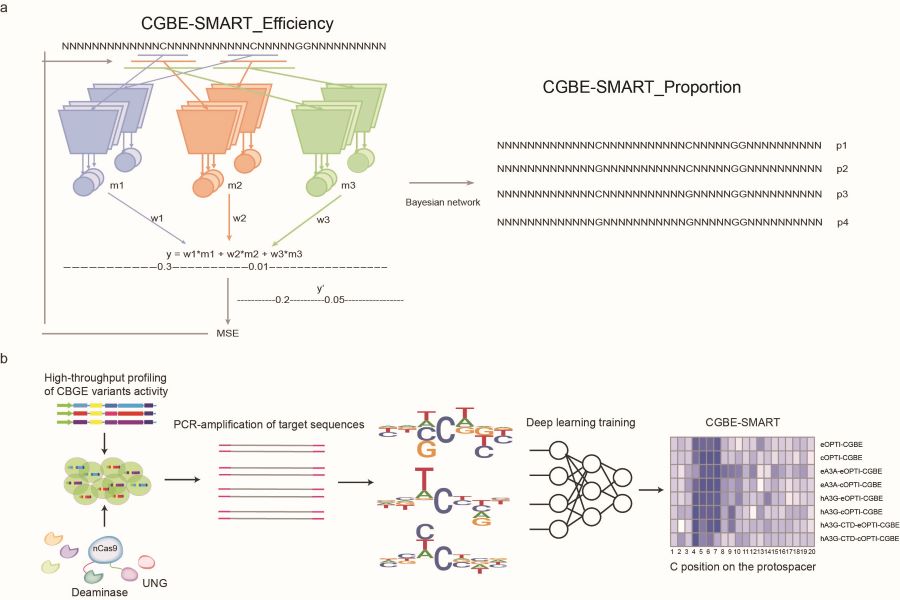
图1. CGBE深度学习模型方法构建思路。a,CGBE-SMART深度学习模型示意图。b,CGBE-SMART预测模型的设计。
总之,研究人员全面优化了CGBE碱基编辑器,获得了高编辑效率与低脱靶的OPTI-CGBEs;并通过机器学习开发了CGBE-SMART深度学习模型用于预测OPTI-CGBEs编辑结果。该研究将进一步加速CGBE的应用研究。
 附件下载:
附件下载: 