

发布时间:2020-05-18
CRISPR/Cas9的衍生工具DNA单碱基编辑技术可以在不切断DNA双链的情况下实现单核苷酸的定向突变,为单碱基突变引起的遗传性疾病的治疗带来了希望,自2016年首次报道以来受到了广泛的关注。2019年,单碱基编辑工具的安全性受到了质疑,先是杨辉等研究组报道了胞嘧啶单碱基编辑器存在严重的DNA脱靶,接着Keith Joung团队、David Liu团队和杨辉团队又分别报道了胞嘧啶单碱基编辑器和腺嘌呤单碱基编辑器存在大量的RNA脱靶效应。虽然先前的研究中通过引入突变的方式显著降低了RNA的脱靶,但是胞嘧啶单碱基编辑器的DNA脱靶依然没有得到解决。
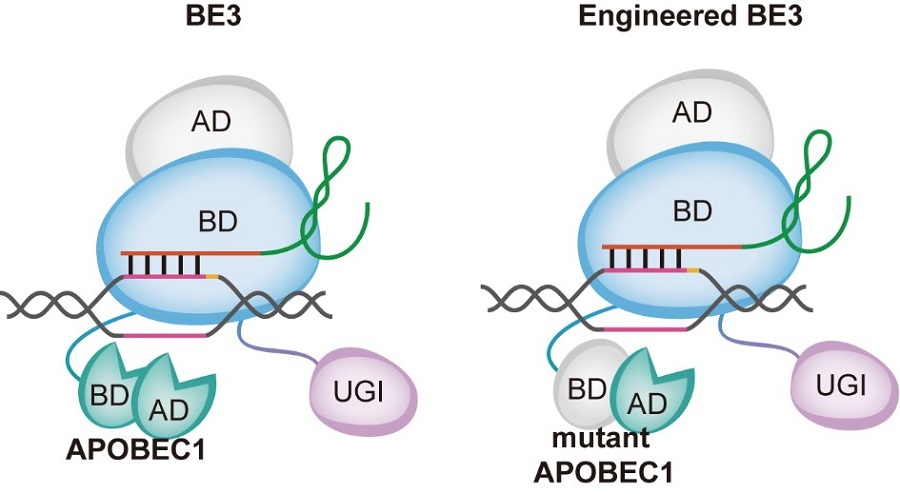
5月18日,《Nature Methods》期刊在线发表了题为《A rationally engineered cytosine base editor retains high on-target activity while reducing both DNA and RNA off-target effects》的研究论文,该研究由中国科学院脑科学与智能技术卓越创新中心(神经科学研究所)杨辉研究组、中国科学院上海营养与健康研究所隶属的计算生物学研究所(中国科学院-马普学会计算生物学研究所)李亦学研究组和中国农业科学院深圳农业基因组研究所左二伟研究组合作完成。该研究根据蛋白结构预测了脱氨酶ssDNA结合的重要氨基酸,在不影响催化活性的情况下,突变相应的氨基酸(APOBEC1上的ssDNA结构域相应氨基酸),从而得到了显著降低DNA脱靶的CBE突变体。
CBE的脱靶是由脱氨酶产生的。脱氨酶利用自身的ssDNA和RNA结合能力,携带Cas9蛋白在基因组或者转录组中随机与ssDNA和RNA结合,并且利用自身催化活性将C突变为T,从而造成单碱基基因编辑工具基因组和转录组范围内完全随机无法预测的脱靶效应。对此,研究者通过两种方式试图降低CBE的脱靶,第一种是在CBE的脱氨酶APOBEC1上引入突变,以此消除ssDNA和RNA的结合能力。他们一共构建了23个CBE突变体,其中 4个突变体不影响基因编辑效率检测,而后通过DNA和RNA的脱靶检测后获得的3个突变体BE3R126E、BE3R132E和YE1-BE3能够显著降低DNA和RNA的脱靶SNV。该方法是通过突变APOBEC1上的核酸结合域关键位点,改变蛋白构象,破坏结合能力,降低随机脱靶。另外一种方式是利用来自于人的APOBEC3A蛋白替换APOBEC1蛋白,并在APOBEC3A的ssDNA结合位点引入突变,但是这种方式只能降低RNA的脱靶,而不能降低DNA上的脱靶。因此,本研究以YE1-BE3为研究重点。为进一步提高YE1-BE3编辑效率,研究者随后又在突变体基础上增加标签和核定位序列(FNLS)。优化后的单碱基编辑工具YE1-BE3-FNLS在保证高保真的情况下,显著提高了基因编辑效率,从而成为既安全又高效的新型基因编辑工具。
该研究结果和David Liu团队今年2月10日发表在《Nature Biotechnology》的研究结论一致,两篇文章都报道YE1在保持较高的编辑效率的同时降低了DNA和RNA上的脱靶,同时缩小了编辑窗口,并降低了indel产生的比例。但是David Liu团队基于细菌抗性筛选的方法只适用于CBE,而杨辉团队基于GOTI的方法是不受限制的,不仅可以检测单碱基编辑器,还可以用于其它基于融合蛋白的基因编辑工具的安全性检测和改进。在本研究中杨辉团队使用GOTI和RNA-Seq同时检测了突变体的DNA脱靶和RNA脱靶,并且发现DNA和RNA的脱靶是互相独立的,需要同时检测。他们获得的 YE1-BE3-FNLS是高精度、高活性单碱基编辑工具,显著降低了脱靶效应,提高了编辑效率,有望应用于遗传疾病基因治疗,推动基因编辑临床化应用。
中国农业科学院深圳农业基因组研究所左二伟研究员,中科院分子细胞卓越创新中心孙怡迪博士,中国农业科学院深圳农业基因组研究所助理研究员袁堂龙,中国科学院脑科学与智能技术卓越创新中心贺冰冰和周昌阳博士为论文共同第一作者,杨辉研究员、李亦学研究员和左二伟研究员为共同通讯作者。
 附件下载:
附件下载: 